Introduction
La Désorption-Ionisation Laser Assistée par Matrice (MALDI) a été mise au point par Michael Karas et Franz Hillenkamp en 1985, afin de répondre au besoin d’analyser des biomolécules de grande taille, notamment des protéines. C’est une ionisation douce permettant de préserver la molécule intacte lors de son ionisation. À l’origine, l’ionisation par laser sur des molécules déposées à l’état solide produisait des ions, mais la sensibilité était très faible.
En 1985, Karas découvre qu’il est possible d’augmenter considérablement la sensibilité en mélangeant l’analyte avec une molécule possédant un noyau aromatique fortement absorbant l’énergie du laser, comme le tryptophane. Cette approche marque la naissance du concept moderne de matrice, permettant une ionisation douce et efficace des molécules de haut poids moléculaire.
En suivant cette même idée, Koichi Tanaka propose une variante utilisant une autre matrice et un laser à 337 nm, au lieu du laser à 266 nm utilisé par Karas. Grâce à cette méthode, Tanaka parvient à ioniser des protéines de grande masse, notamment la carboxypeptidase (34 472 Da). De son côté, Karas réussit également à ioniser des protéines encore plus massives, comme l’albumine (67 kDa), avec un laser à 266 nm.
En raison de l’importance majeure de cette source d’ionisation, le Prix Nobel de chimie 2002 a été attribué à Koichi Tanaka. Cette attribution a suscité de nombreuses controverses, car une grande partie de la communauté scientifique estimait que le prix aurait dû revenir à Karas et Hillenkamp, considérés comme les véritables fondateurs du MALDI moderne.
Principe du MALDI
L’ionisation par MALDI se déroule sous vide, dans la source du spectromètre de masse. Un laser pulsé vient irradier le cristal matrice/analyte (figure 1). Bien que le mécanisme exact ne soit pas entièrement élucidé, plusieurs phénomènes clés sont généralement admis :
-
la matrice absorbe fortement l’énergie du laser ;
-
cette absorption provoque une élévation rapide de la température ;
-
le chauffage brutal entraîne une expansion explosive du cristal, suivie d’une désorption du mélange sous forme de panache gazeux ;
-
au cours de cette expansion, les molécules d’analyte sont ionisées.
Plusieurs mécanismes d’ionisation sont proposés, notamment :
-
la photoionisation en phase gazeuse,
-
le transfert de proton entre la matrice et l’analyte,
-
la désorption d’ions préformés.
Une fois formés, les ions sont accélérés par un champ électrostatique vers l’analyseur du spectromètre.
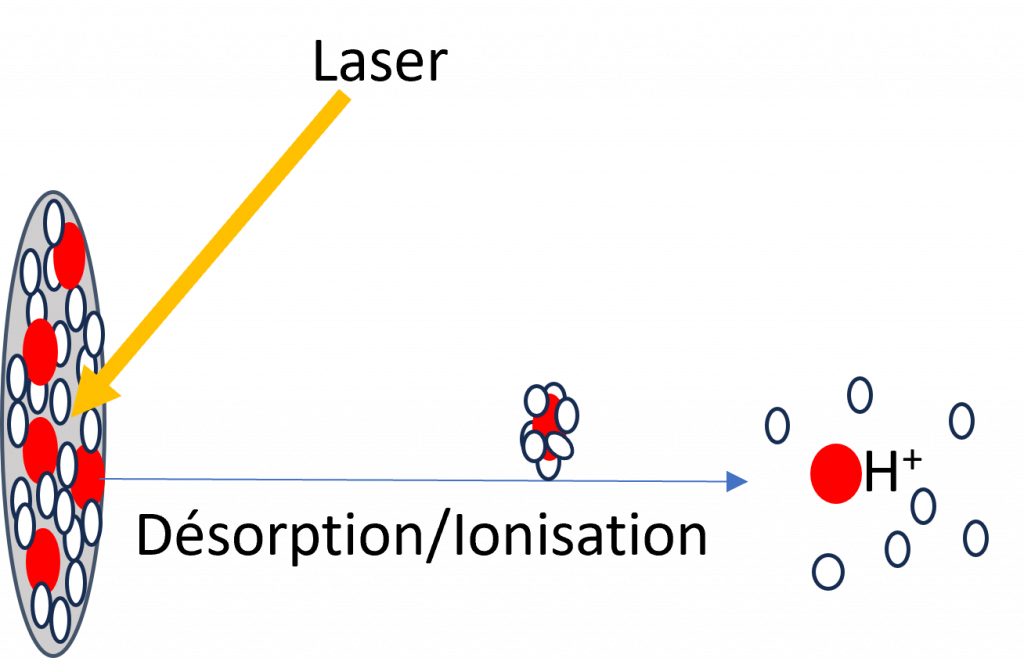
Les ions produits par MALDI sont générés sous forme de paquets d’ions, c’est-à-dire qu’ils sont formés simultanément à la suite de chaque impulsion laser. Pour cette raison, l’ionisation MALDI est très fréquemment couplée à un analyseur à temps de vol (Time-of-Flight, TOF), particulièrement bien adapté à ce mode de production pulsé. L’analyseur TOF reçoit le paquet d’ions et les sépare en fonction de leur rapport masse/charge (m/z), les ions les plus légers atteignant le détecteur en premier.
Il est également possible de coupler une source MALDI avec d’autres analyseurs capables d’accepter des paquets d’ions, tels que l’Orbitrap, le FT-ICR ou les pièges ioniques. En revanche, le couplage direct de MALDI avec un quadripôle est en pratique très limité, car ce dernier fonctionne en régime continu. Il n’est envisageable que dans des modes ciblés spécifiques, et, à notre connaissance, ce type de configuration reste extrêmement rare, voire inexistant dans les instruments commerciaux courants.
Depuis l’invention du MALDI, un grand nombre de matrices ont été développées, permettant l’analyse de molécules de très haut poids moléculaire, pouvant atteindre 300 kDa, voire davantage, ce qui constitue l’un des atouts majeurs de cette technique.
Limites du MALDI
Le MALDI présente toutefois plusieurs inconvénients :
1. Les ions monochargés
Les ions formés sont essentiellement monochargés
Conséquences :
-
Des valeurs de m/z très élevées, ce qui explique pourquoi la source MALDI est le plus souvent couplée à un analyseur à temps de vol (TOF). En effet, contrairement à la majorité des autres analyseurs, le TOF conserve une résolution relativement élevée pour les ions de très grand m/z, la séparation reposant sur le temps de vol plutôt que sur un confinement ou une oscillation dépendant fortement de la masse.
-
une gamme de masse très large, rendant l’analyse plus complexe.
Ainsi, un même spectre MALDI peut contenir des ions allant de quelques centaines de daltons jusqu’à plusieurs dizaines, voire centaines, de kilodaltons.
2. Difficulté de coupler avec les méthodes séparatives
Le MALDI est difficile à coupler en ligne (online) avec la LC.
Un couplage off-line est possible, mais l’optimisation du rapport matrice/analyte devient alors complexe, ce qui peut affecter la sensibilité et la reproductibilité.
Préparation des échantillons en MALDI
La préparation des échantillons est une étape cruciale en MALDI, car elle conditionne directement la qualité de l’ionisation et la sensibilité de l’analyse. Elle consiste à mélanger l’analyte avec une matrice, c’est-à-dire une petite molécule organique capable d’absorber efficacement l’énergie du laser.
Différentes matrices ont été développées selon le type de molécules à analyser :
-
HCCA (α-cyano-4-hydroxycinnamic acid) : adaptée aux peptides, aux petites molécules et à certains sucres.
-
SA (acide sinapinique) : particulièrement efficace pour les protéines.
-
THAP (2,4,6-trihydroxyacétophénone) : utilisée pour les oligonucléotides de petite taille.
-
Acide nicotinique : recommandé pour les oligonucléotides de plus grande taille.
Une fois la matrice sélectionnée, il est également essentiel de déterminer le meilleur ratio matrice/analyte, car un rapport inadapté peut entraîner une mauvaise cristallisation ou une ionisation inefficace. L’optimisation de ce ratio permet d’obtenir des cristaux homogènes et assure une analyse MALDI optimale.
Imagerie MALDI
Une application particulièrement intéressante du MALDI est l’imagerie MALDI (MALDI-MSI : MALDI Mass
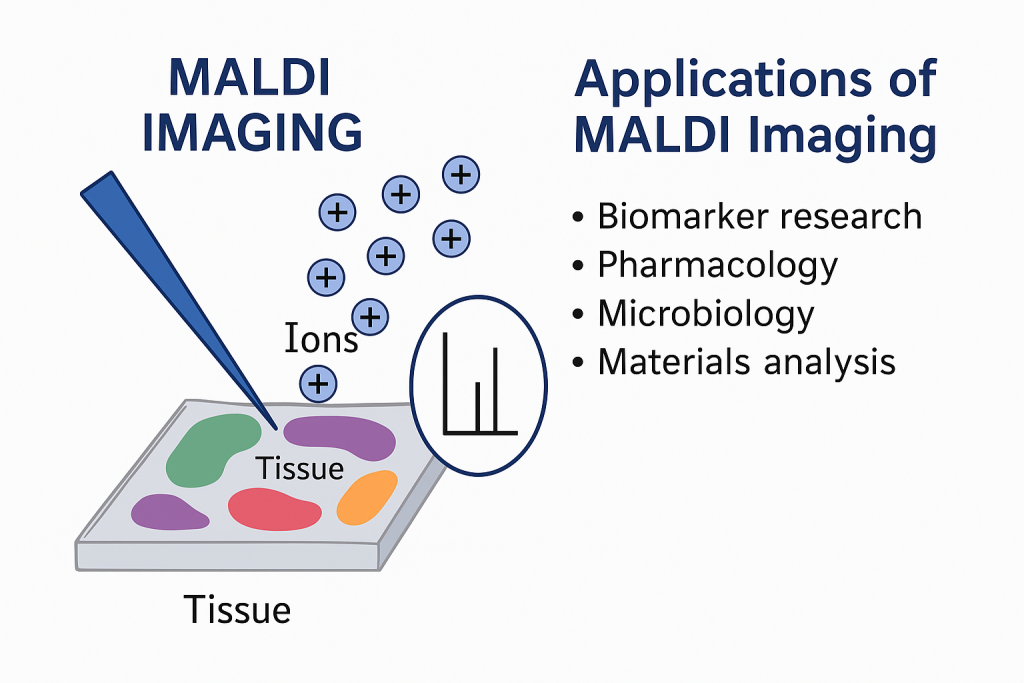
Spectrometry Imaging). Il s’agit d’une technique analytique innovante qui permet de cartographier la distribution spatiale des molécules directement à la surface d’un tissu biologique, d’une préparation histologique ou d’un matériau. Comme le rayon laser ne mesure que quelques dizaines de micromètres, il est possible, en déplaçant le point d’impact du laser sur différents emplacements d’un tissu pouvant mesurer plusieurs millimètres, de reconstituer une carte moléculaire. En suivant l’intensité d’un ion particulier, on peut ainsi visualiser la distribution locale et la concentration relative de la molécule dans l’échantillon. L’imagerie MALDI combine donc la spectrométrie de masse et l’imagerie spatiale, fournissant simultanément des informations moléculaires, spatiales et semi-quantitatives. Elle est couramment utilisée pour la cartographie des lipides, métabolites, peptides, protéines, et bien d’autres classes de biomolécules.
